ProteoformX™是一款功能强大且用户友好的软件平台,专为简化完整蛋白质及 Top-Down 质谱分析而设计。平台结合快速完整蛋白质质量分析功能,全局评估可检测的proteoform的多样性,同时提供深入的Top-Down 分析,能够解析完整蛋白质序列、精准定位翻译后修饰位点,并实现proteoform的定量分析。
Key Features
Intact Mass 分析

Top-down Proteoform 搜索
Proteoform Manager
Intact Mass 分析
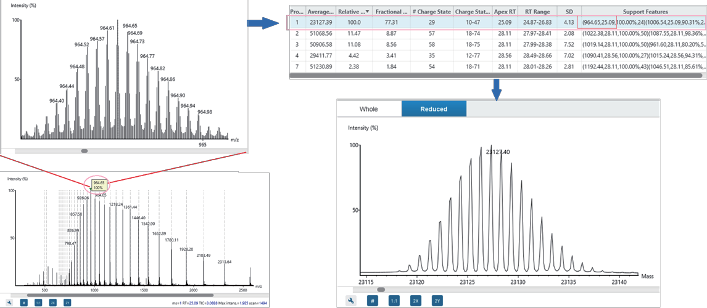
高精度质谱解卷积
基于特征峰的质量解卷积算法能够以高灵敏度和特异性识别proteoform的质量,适用于同位素区分的高分辨和低分辨的数据,可处理广泛的样本类型。
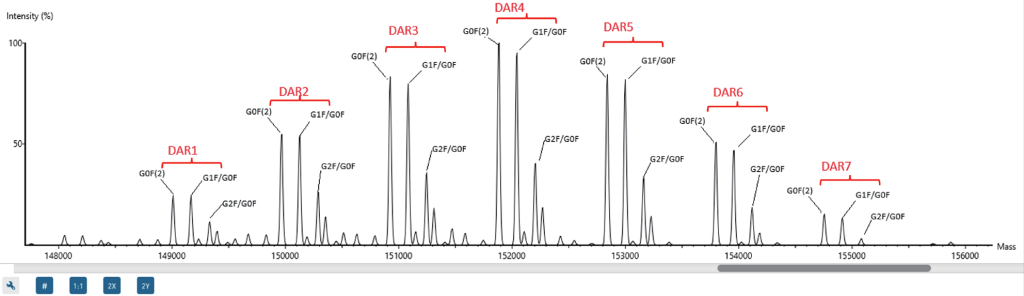
带修饰和ADC药物有效载荷样本的全面注释
完整分子量分析工作流可为proteoform复合体和亚基提供准确注释,即使是带有修饰和 ADC 药物有效载荷的高度异质性的样本也同样适用。
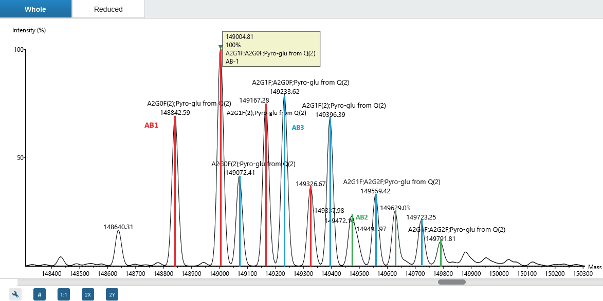
Proteoform水平上的糖基化分析
完整分子量解卷积技术可精确检测抗体混合物中的糖基化proteoform。
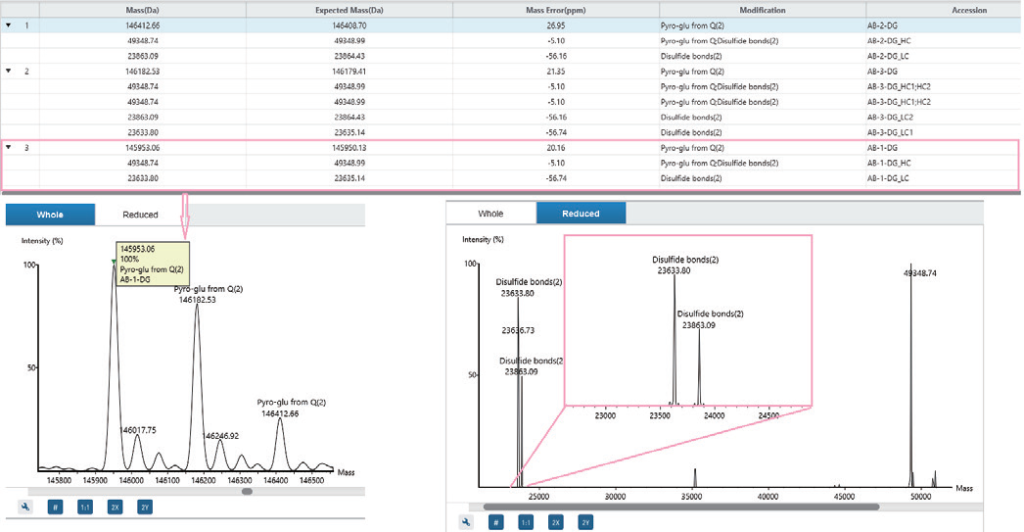
抗体重链与轻链配对分析
完整分子量蛋白质注释可确定抗体重链与轻链的配对方式,从而对所有抗体形式(包括不对称抗体和混合物)进行全
面表征。
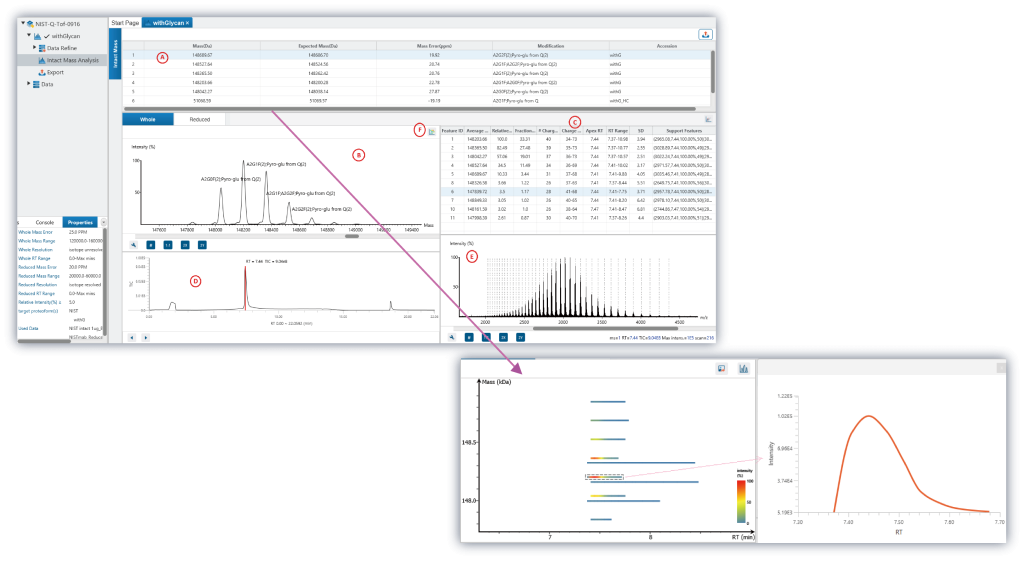
全面的结果可视化
ProteoformX™ 提供全面的分析结果,包括带有注释峰的交互式谱图视图以便用于透明化解析,以及解卷积得到的所有质量数表格中 support feature 信息,可直接用于指导靶向top-down的MS/MS实验。
Top-down蛋白质组搜索
Top-down质谱技术可实现Proteoform的表征,包括精确的翻译后修饰定位。然而,数据复杂性和序列覆盖度有限等挑战可能会限制分析和通量。在ProteoformX™中,其用户界面可帮助用户对从原始数据到鉴定结果的全流程进行手动核查。ProteoformX支持多种碎裂方法(CID、HCD、ETD、ETHCD,UVPD)。
![]() Bottom-up辅助的Top-down/middle-down质谱分析
Bottom-up辅助的Top-down/middle-down质谱分析
![]() 通过分层级追踪每一种proteoform
通过分层级追踪每一种proteoform
![]() 支持多种碎裂方法
支持多种碎裂方法
![]() 开放搜索
开放搜索

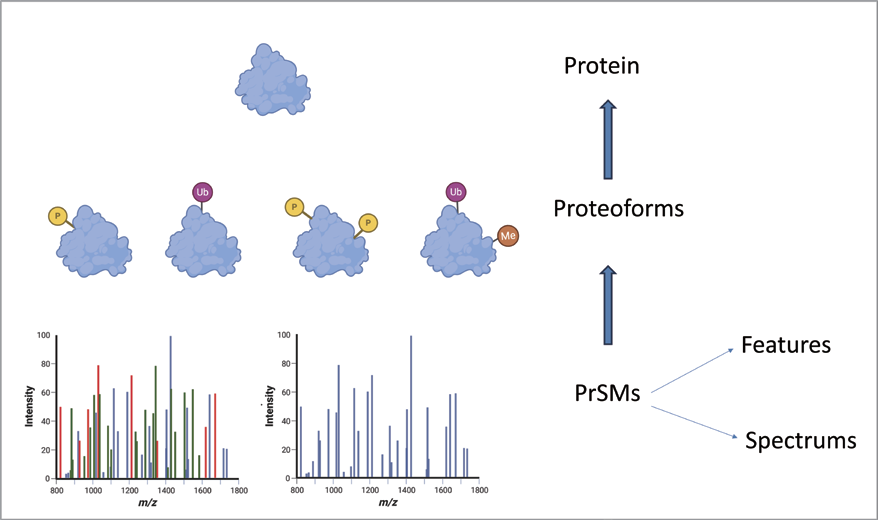
ProteoformX™ 软件通过基于特征峰的识别算法提供分层信息。
具有灵活的用户交互界面,可实现不同视图之间的链接,从而能够让用户无缝获取从原始数据到蛋白质形态谱匹配(PrSM)及蛋白质推断的信息。
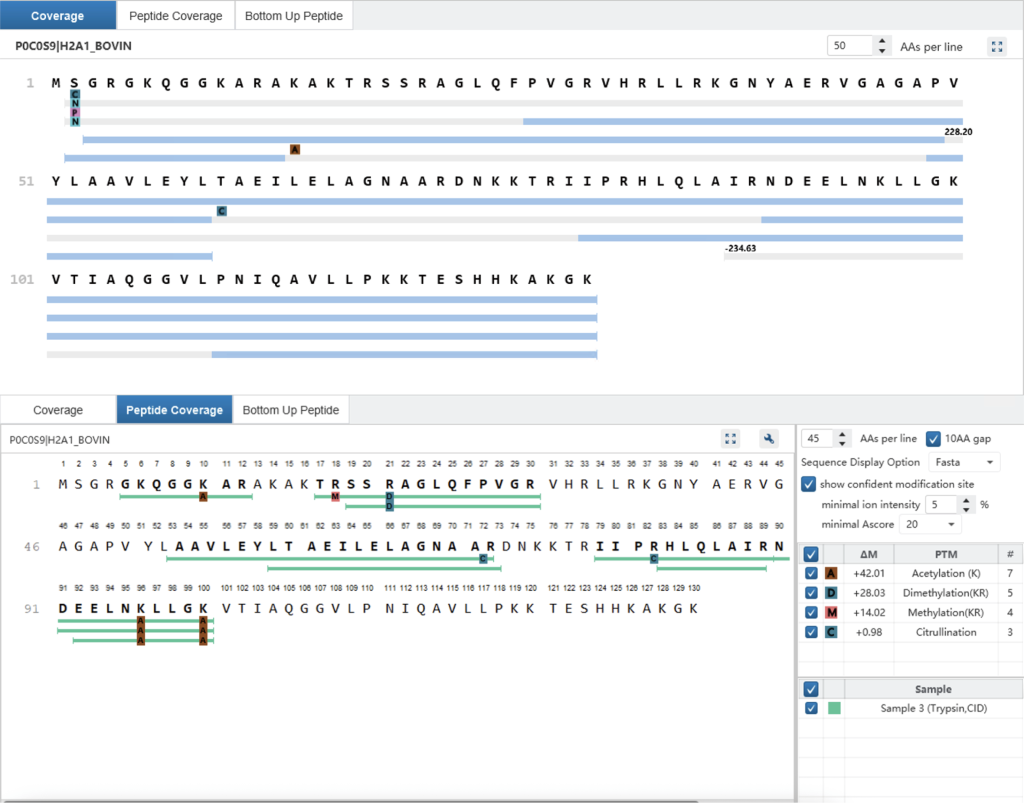
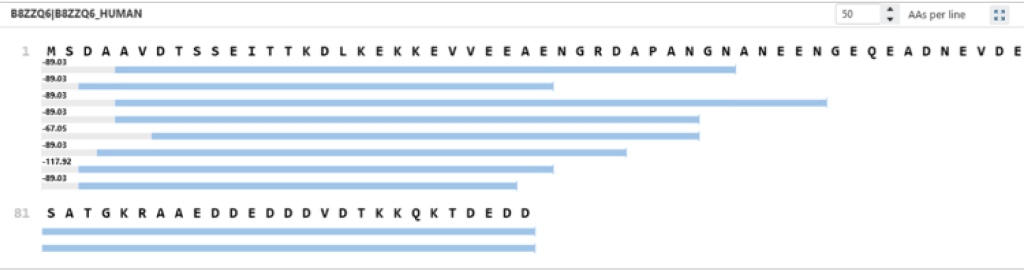
在Protein tab,中,覆盖度面板展示了映射至蛋白质序列上的 proteoform,而肽段覆盖度则显示所有来自 bottom-up质谱分析所匹配到该蛋白质序列的所有肽段。在覆盖度视图中,灰色条带代表已匹配至蛋白质序列的区域,其对应的质量偏移暗示可能存在修饰。但这些区域缺乏用于质量标签位点特异性定位的碎片离子。
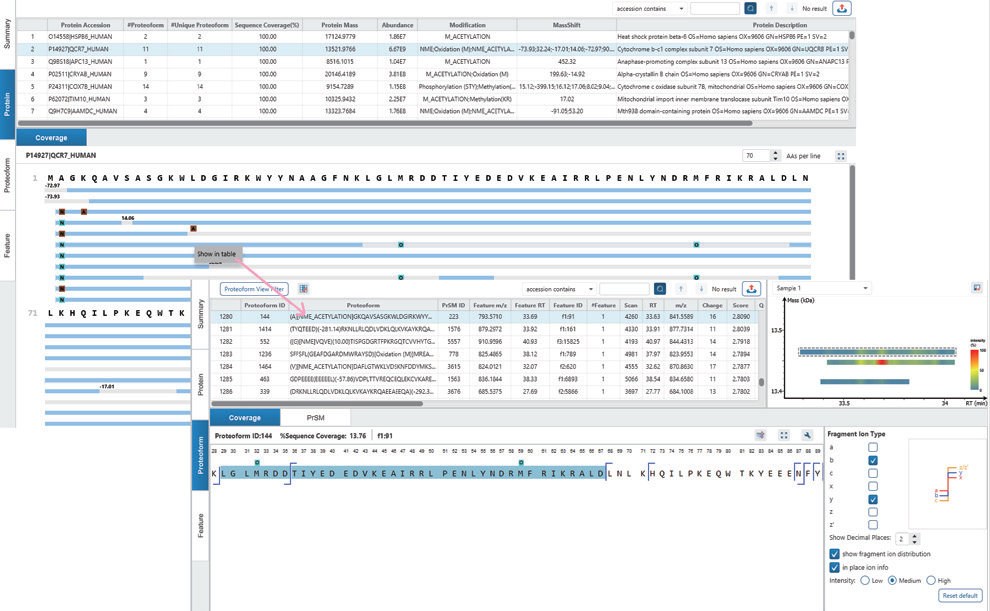
在 Proteoform tab中, 序列覆盖度由top-down碎片离子支持的氨基酸序列百分比来定义。相对于氨基酸序列质量的修饰或是质量偏移会在结果视图中呈现。若某修饰可通过bottom-up数据定位到单个氨基酸,top-down碎片中对应的残基将标黄突出显示(图 8)。
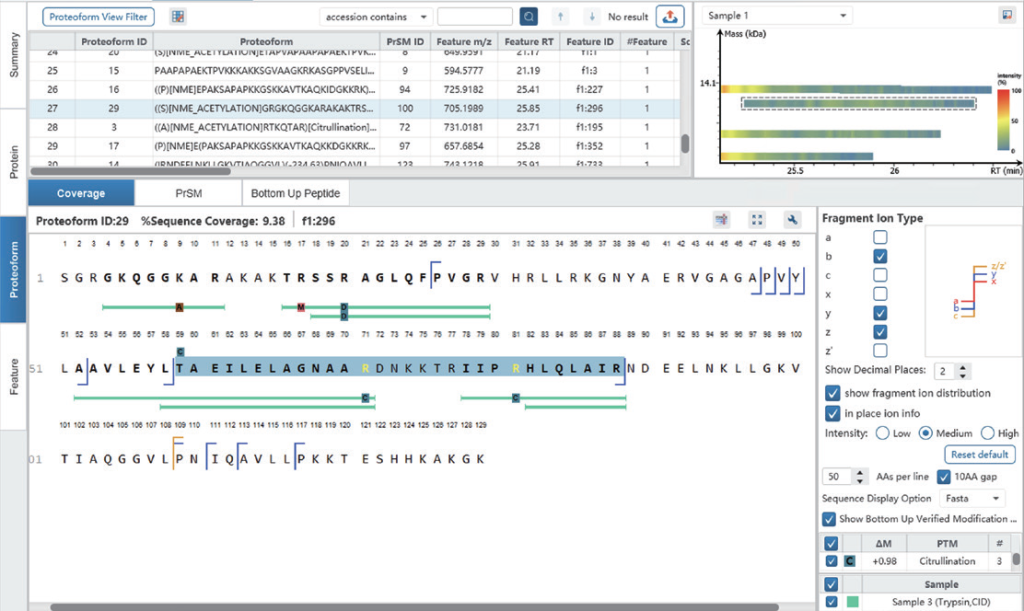
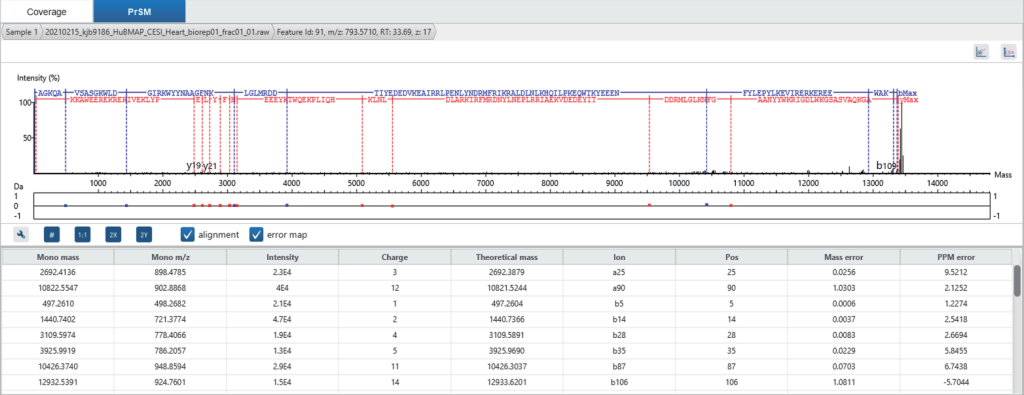
深入查看 Proteoform tab,可以看到每个proteoform鉴定结果是如何以高质量PrSMs(Proteoform Spectrummatch)、碎片离子质量表和交互式序列碎裂图作为支撑的。ProteoformX™的“Spectrum tab”将理论母离子峰和碎片离子分别直接叠加到MS1和MS2谱图上,可直观看到同位素峰簇分布,评估离子隔离窗口,精准定位参与解卷积的离子,获取哪些谱是来自嵌合谱图的信息。不同颜色和形状注释清晰区分离子类型,匹配与未匹配碎片,为结果提供透明度与可信度。
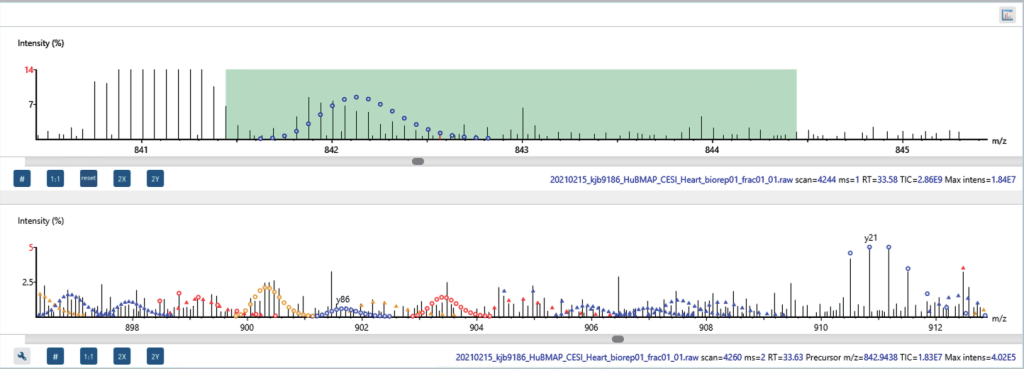
基于特征峰的数据洞察(LC-MS视图)
作为用于完整蛋白质和top-down分析的基于特征峰的算法,ProteoformX™ 提供包含 ms/ms 谱图及其相关特征峰的LC-MS视图。可放大以详细查看每个特征峰。
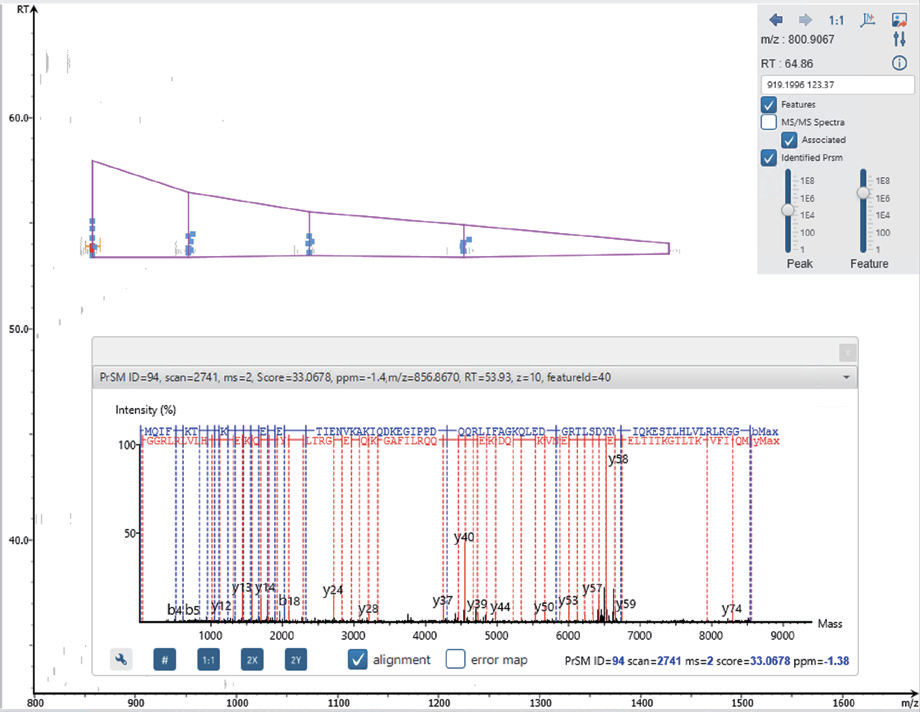
基于开放搜索的深度自上而下蛋白质组鉴定
通过启用OpenPTM搜索功能,允许检测预期以外的修饰和蛋白质的截断形式,从而提升Proteoform的鉴定能力。在以下示例中,未启用OpenPTM搜索时仅鉴定出B8ZZQ6蛋白的2种C端Proteoform,而启用后可检测到8种。其中新增的一种Proteoform呈现‒89.03 Da的质量偏移,这与N端蛋氨酸缺失伴随乙酰化修饰(一种常在Protein N端被观察到的修饰)的特征一致。通过扩展搜索空间以包含质量偏移和非规范末端[non-canonical termini],OpenPTM搜索可实现更全面的Proteoform分析。

Proteoform Manager
蛋白质异构体的灵活定义与可视化呈现
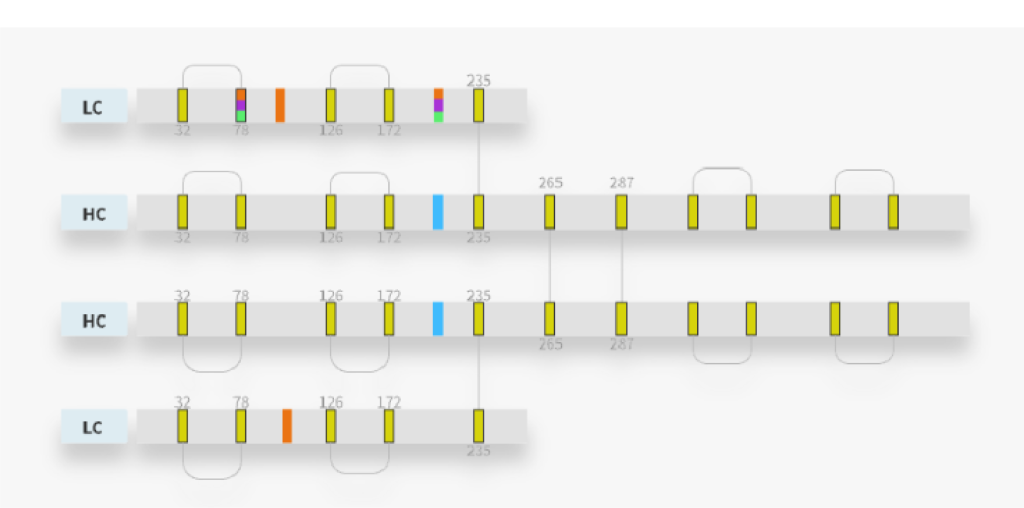
ProteoformX™ 中的 Proteoform Manager 提供了直观的界面,可用于定义链内与链间连接、切割位点、位点特异性翻译后修饰(PTM)以及糖基化。这些功能支持对生物制药分析相关的复杂proteoform进行全面建模。
通过模块化界面,用户可交互式自定义蛋白质。可视化编辑器采用彩色编码符号和直观注释,突出显示序列修饰和结构变异,方便用户轻松构建、修改并验证含突变或异质性糖型的proteoform。
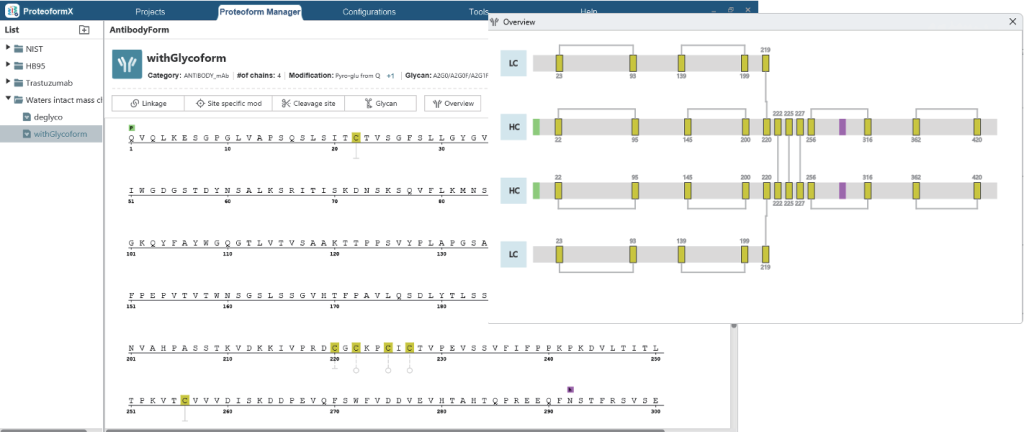
例如,轻松使用的Proteoform Manager在Proteoform水平定义抗体,包括位点特异性修饰、糖基化形式、连接和切割位点。
References & Resources
1.Roberts, D.S., Loo, J.A., Tsybin, Y.O. et al. Top-down proteomics. Nat Rev Methods Primers 4, 38 (2024). https://doi.org/10.1038/s43586-024-00318-2



