深入糖蛋白组学和糖组学分析的高级解决方案

联系我们咨询全新的 PEAKS GlycanFinder
核心功能
仪器商中立的分析软件
为实现最佳的糖肽鉴定性能,GlycanFinder 整合了通过不同碎裂方式数据集训练的优化算法。软件可支持多种质谱设备。

增强聚糖结构解析可信度
GlycanFinder 采用基于结构的糖肽鉴定方式。S-Score (%)用于表征所选糖肽结构相较于其他聚糖同分异构体候选物的支持程度。

用户友好的操作界面
PEAKS GlycanFinder 提供了直观易用的操作界面,显著简化了糖链分析流程。用户可通过交互式、信息丰富的可视化查看器深入探索分析结果,并能便捷地导出相关数据,用于团队共享或下游生物信息学分析。

多个O糖基化位点分析
GlycanFinder 3.0 的完整糖肽数据库搜索现已支持自定义每条糖肽上O糖的位点数,最多可设置5个,实现准确的复杂糖肽鉴定和糖基化位点定位。尽管组合搜索空间显著扩大,GlycanFinder 3.0通过应用高效过滤算法、位点特异性定位评分以及基于结构的匹配策略,在保持快速分析性能的同时,仍能提供高置信度的糖基化位点鉴定结果。

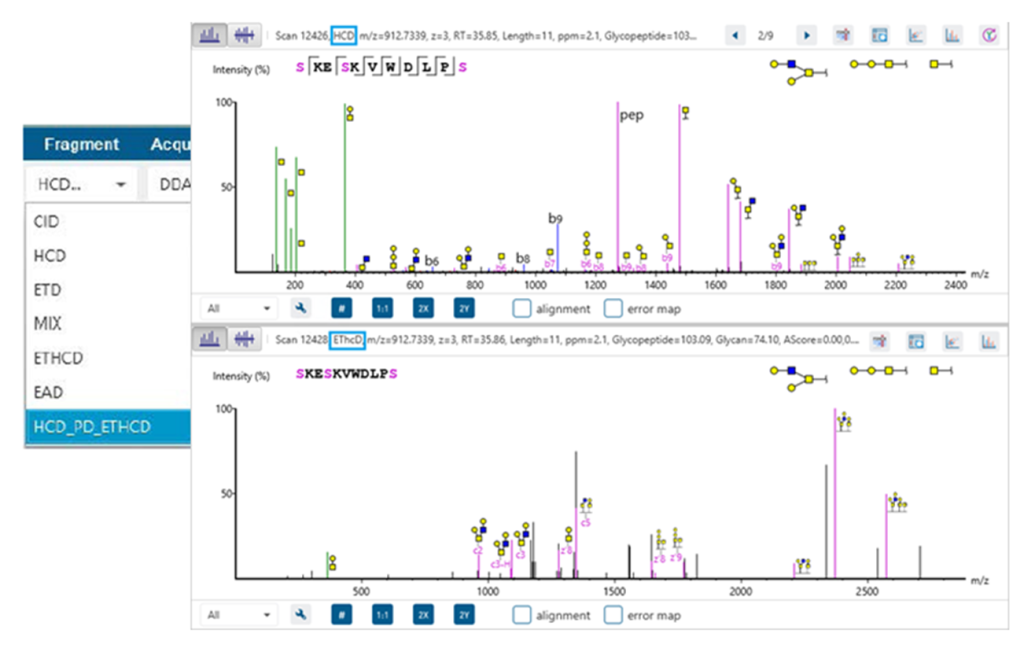
配对谱图支持
新增HCD-pd-EThcD混合谱数据分析,可实现糖链结构与糖基化位点的高置信度鉴定。GlycanFinder 3.0可分别显示同一条糖肽的HCD和EThcD的注释谱图,便于用户直观解析鉴定结果。
聚糖从头测序
当糖肽谱图无法与现有数据库匹配时,PEAKS GlycanFinder 可通过 N – 糖链从头测序技术,直接从谱图中推断糖链结构。这一功能能够助力发现已配置聚糖数据库中未包含的额外糖链结构。

聚糖修饰分析
PEAKS® GlycanFinder 提供全面的糖组学分析方案,用于表征具有多种修饰的糖肽以及游离糖,以解决聚糖修饰带来的分析挑战。


高重现性的价态匹配
价态匹配通过整合所有前体特征(包括未获得MS2鉴定的特征)来提升分析的重现性。肽段通常会产生多种电荷态,若仅依赖已鉴定的前体进行糖链分析,可能导致结果偏差。价态匹配能够检测到更多相关前体,并利用组合特征峰面积进行谱图分析。

定制聚糖数据库
糖链数据库编辑器通过直观的用户界面,使用户能够构建自定义糖链数据库。用户可通过结构绘制工具添加、编辑和修改糖链结构,并轻松将其导入软件中。

InChorus – 跨软件结果比对
在糖蛋白质组学研究中,不同分析软件对糖肽的鉴定结果往往存在差异,这会导致注释结果不一致或模糊不清。GlycanFinder 3.0全新的InChorus功能可以整合多个搜索软件的糖肽鉴定结果,在用户友好的界面中清晰展示共有和各自的糖肽鉴定结果。InChorus可自动提取其他搜库软件的数据,在单张谱水平上直观展示每个PSM的重叠情况,并提供直观的筛选和导出选项,用户能够更清晰地获得更高置信度的分析结果。

聚糖网络
Glycan Network展示了各种糖型之间的关联,每条边连接的两个糖之间仅有一个糖单元组成不同,因此可将具有共同核心结构的、末端不同的糖全面可视化。使用户能够清晰观察各糖链从共同核心结构向外延伸的糖基化分支模式。

高灵敏度分析
升级的评分算法通过整合碎片离子强度、聚糖特异性特征离子、肽段骨架离子及互补碎裂模式,显著提升了分析灵敏度。这一优化改善了低信号谱图的检测能力,实现了糖肽的高可靠性鉴定。

References
- Pinho, S., Reis, C. Glycosylation in cancer: mechanisms and clinical implications. Nat Rev Cancer 15, 540–555 (2015). doi:10.1038/nrc3982
- Sun, W., Zhang, Q., Zhang, X. et al. Glycopeptide database search and de novo sequencing with PEAKS GlycanFinder enable highly sensitive glycoproteomics. Nat Commun 14, 4046 (2023). doi:10.1038/s41467-023-39699-5
- Korkola, N., Jurcic, K. PEAKS GlycanFinder 2.5: Launching a New Era for Glycoproteomics and Glycomics. Bioinformatics Solutions Inc (2025). https://www.bioinfor.com/peaks-glycanfinder-2-5-launching-a-new-era-for-glycoproteomics-and-glycomics